Le programme de recherche ATLASea fait partie des lauréats des PEPR (Projets et Équipements Prioritaires de Recherche) exploratoires, qui visent des secteurs scientifiques ou technologiques en émergence pour lesquels l’État souhaite identifier et structurer ces communautés. Co-piloté par le CNRS et le CEA, ce programme est financé par France 2030 sur 8 ans. Il a pour but de séquencer les génomes de 4500 espèces marines eucaryotes (espèces dont les cellules sont dotées d’un noyau) comprenant : mollusques, crustacés, annélides, cnidaires, ascidies, algues unicellulaires et pluricellulaires, éponges et poissons, soit environ un tiers des espèces marines connues de l’hexagone et des territoires ultramarins. Les données récoltées seront déposées dans une base de données en libre accès pour la communauté scientifique et compléteront ainsi les inventaires de biodiversité. Sous l’égide du Earth Biogenome Project (EBP), il existe plus de 50 programmes internationaux qui ont pour but d’obtenir des génomes de référence.
Pourquoi séquencer les génomes d’espèces marines ?
L’avancée phénoménale des technologies de séquençage au cours des deux dernières décennies ouvre aujourd’hui des possibilités sans précédent pour comprendre le vivant. De plus, on estime qu’il existe 8,7 millions d’espèces eucaryotes dans le monde, dont environ 2,2 millions d’espèces marines. Les efforts de ciblage sur la microfaune et l’analyse moléculaire pourraient augmenter le nombre d’espèces identifiées, d’autant que le monde marin compte le plus grand nombre de lignées exclusives, c’est-à-dire d’organismes n’ayant jamais quitté l’océan. Aussi, cette diversité d’espèces se traduit par des modes d’existence et d’adaptation à l’environnement tout à fait remarquables et largement sous-étudiés. C’est pourquoi ATLASea ambitionne de produire un socle de données génomiques de référence sur les organismes marins de la zone économique exclusive française.
Les organismes sont liés par leur ADN, hérité d’ancêtres commun depuis l’origine de la vie. Ce génome contient, pour chaque organisme, l’ensemble des instructions nécessaires au développement de l’organisme et au fonctionnement de l’adulte. Ces données permettront de mieux comprendre les mécanismes moléculaires qui sous-tendent la physiologie de ces espèces, et de retracer leur histoire évolutive à travers leurs génomes. En effet, analyser les génomes d’espèces modernes est un moyen efficace de remonter le temps, et d’accéder à des informations génétiques sur leurs ancêtres depuis longtemps disparus. Le séquençage de milliers d’espèces eucaryotes est donc un outil puissant pour retracer cette histoire et mieux comprendre les origines du vivant. Par ailleurs, mieux connaître la biodiversité marine ainsi que sa dynamique aidera à mieux préserver les milieux marins. Enfin, la découverte de voies métaboliques menant à de nouvelles molécules d’intérêt sera utile à la santé, l’agriculture ou l’alimentation.
Les étapes du projet ATLASea
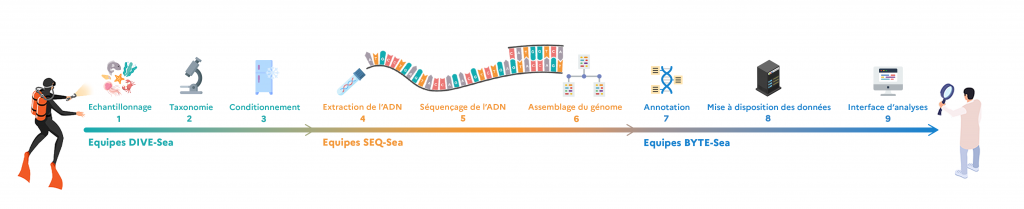
Pour obtenir les résultats attendus, marins, plongeurs et chercheurs vont d’abord devoir récolter les espèces marines sur une dizaine de sites répartis le long du littoral français et en Outre-mer (Nouvelle-Calédonie, Guadeloupe, Mayotte Polynésie française), couvrant diverses conditions climatiques, géomorphologiques et environnementales. Un large éventail de méthodologies de collecte sera déployé pour maximiser la diversité des espèces et des groupes taxonomiques prélevés : Les bateaux déploient du matériel de prélèvement de différentes tailles et adaptés à différentes profondeurs ou types de fonds. Les plongeurs utiliseront des flacons avec capuchon à vis pour la récolte à vue, aspirateurs marins pour prélever à la surface des rochers et les fonds meubles, paniers et brosses pour récupérer la faune qui se cache sous les blocs rocheux. Les tissus des mammifères marins seront prélevés uniquement à l’occasion d’échouages se produisant naturellement. En parallèle, des espèces seront cultivées dans les stations marines de Roscoff, Banyuls ou Villefranche-sur-Mer.
Une fois prélevés, les spécimens sont identifiés et classés par des taxonomistes aguerris du Muséum national d’Histoire naturelle (MNHN) qui vont vérifier leur statut de séquençage dans les bases de données internationales. S’ils n’y sont pas déjà déclarés, l’étape suivante consiste à rapidement plonger les spécimens dans de l’azote liquide à – 196 °C, afin d’être congelés instantanément, puis préservés dans une épaisse boite de polystyrène, remplie de carboglaces à – 80 °C.
Direction désormais le Génoscope – Centre National de Séquençage. Pendant le transfert, la chaîne du froid doit être rigoureusement respectée, car le congélation des tissus permet d’empêcher la dégradation de l’ADN. Les ingénieurs biologistes du CEA récupèrent ainsi les échantillons et peuvent commencer l’extraction de l’ADN des tissus. Une tâche ardue car l’ADN de certains organismes comme les algues sont difficiles à extraire. Il faut alors mettre en place de nouveaux protocoles de purification pour obtenir de hautes concentrations d’ADN. Avant de se lancer dans le séquençage complet du génome, un séquençage « léger » intervient pour confirmer l’identification de l’espèce, surtout si l’identification seulement sur la base de la morphologie du spécimen ne suffisait pas. Vient ensuite l’étape du séquençage du génome, réalisée par différentes techniques selon les informations requises (Illumina, Pacific Bioscience, Oxford Nanopore Technologies). Les techniques de séquençage ne sont pas capables de séquencer les chromosomes entièrement, elles sont d’abord obligées de les fragmenter en plus ou moins grands morceaux. Mais les méthodes de dernière génération sont heureusement capables de séquencer de très longs fragments à un haut débit, ce qui permet ensuite de les réassembler pour former un génome entier.
Après l’étape de l’assemblage, le génome peut être analysé : c’est la phase d’annotation, qui consiste à extraire ses caractéristiques comme la localisation des gènes, leurs structures, tailles, et fonctions, à partir d’outils informatiques. Pour cela, les bioinformaticiens en charge vont mettre en œuvre des méthodes d’analyse automatisée pour annoter plus rapidement les génomes. Toutes les informations des génomes seront stockées à l’Institut Français de Bio-informatique et seront accessibles sur un portail qui proposera des outils de visualisation pour faire de la génomique comparative ou encore de la reconstruction de génomes ancestraux. Parallèlement, le MNHN conservera les spécimens de référence, sur lesquels auront été prélevés les tissus, à – 80˚C, constituant ainsi des collections naturalistes du futur.
Nos engagements : environnement et science ouverte
Les campagnes d’échantillonnage sont dimensionnées pour laisser l’empreinte la plus faible possible sur les milieux visités. Les plongeurs, qui opèrent la sélection la plus fine en comparaison d’une drague ou d’un chalut, évaluent en temps réel l’opportunité de collecte et le calcul coût/bénéfice d’un prélèvement. Par ailleurs, ATLASea respecte les réglementations en vigueur sur les espèces et les espaces protégés, sur les méthodes de collecte conventionnelles, sur l’utilisation d’animaux à des fins scientifiques. La pratique scientifique en matière de gestion des données s’inscrit dans une démarche FAIR (faciles à trouver, accessibles, interopérables et réutilisables). La cryoconservation a également une empreinte carbone et le nombre d’échantillons conservés à -80 °C sera limité au strict nécessaire. Enfin, ATLASea sera en conformité avec le protocole de Nagoya sur l’accès aux ressources génétiques et le partage juste et équitable des avantages découlant de leur utilisation.
Déclaration de conformité réglementaire, d’éthique et de bonnes pratiques
Les partenaires du programme ATLASea sont responsables du respect de toutes les lois, licences, autorisations, permis et réglementations applicables (tant au niveau national qu’international) relatives à l’acquisition, au transfert, au stockage, à l’utilisation, à la destruction et à l’élimination (le cas échéant) des échantillons ou spécimens qu’ils collectent, et de la mise à disposition des preuves documentaires de ce respect au Comité de Conformité Réglementaire d’ATLASea. Pour toutes ces activités, les partenaires d’ATLASea doivent mettre en œuvre les Procédures Opérationnelles Standard (SOP) d’ATLASea, qui doivent inclure les détails des exigences réglementaires et éthiques. Les collaborateurs qui ne sont pas employés par l’un des partenaires d’ATLASea (voir le schéma de gouvernance) doivent informer leurs représentants légaux qui devront certifier que les conditions énoncées dans la « Déclaration de conformité réglementaire, d’éthique et de bonnes pratiques » seront respectées et en particulier celles énoncées ci-dessus. Les normes décrites dans ce document doivent être maintenues pendant le transfert d’échantillons ou de spécimens de/vers les partenaires de l’équipe ATLASea et tout autre collaborateur.
Vous pouvez consulter le texte de la « Déclaration de conformité réglementaire, d’éthique et de bonnes pratiques » du PEPR ATLASea ici (texte en anglais)
